SimpleFold: تا کردن پروتئینها سادهتر از آن است که فکر میکنید
پژوهشگران بهتازگی از SimpleFold، یک رویکرد انقلابی برای تاخوردگی پروتئین رونمایی کردهاند که روشهای مرسوم را به چالش میکشد. این مدل جدید نشان میدهد که پیشبینی ساختارهای پیچیده سهبعدی پروتئینها ممکن است به آن معماریهای فوقتخصصی و پیچیدهای که تصور میکردیم، نیاز نداشته باشد.
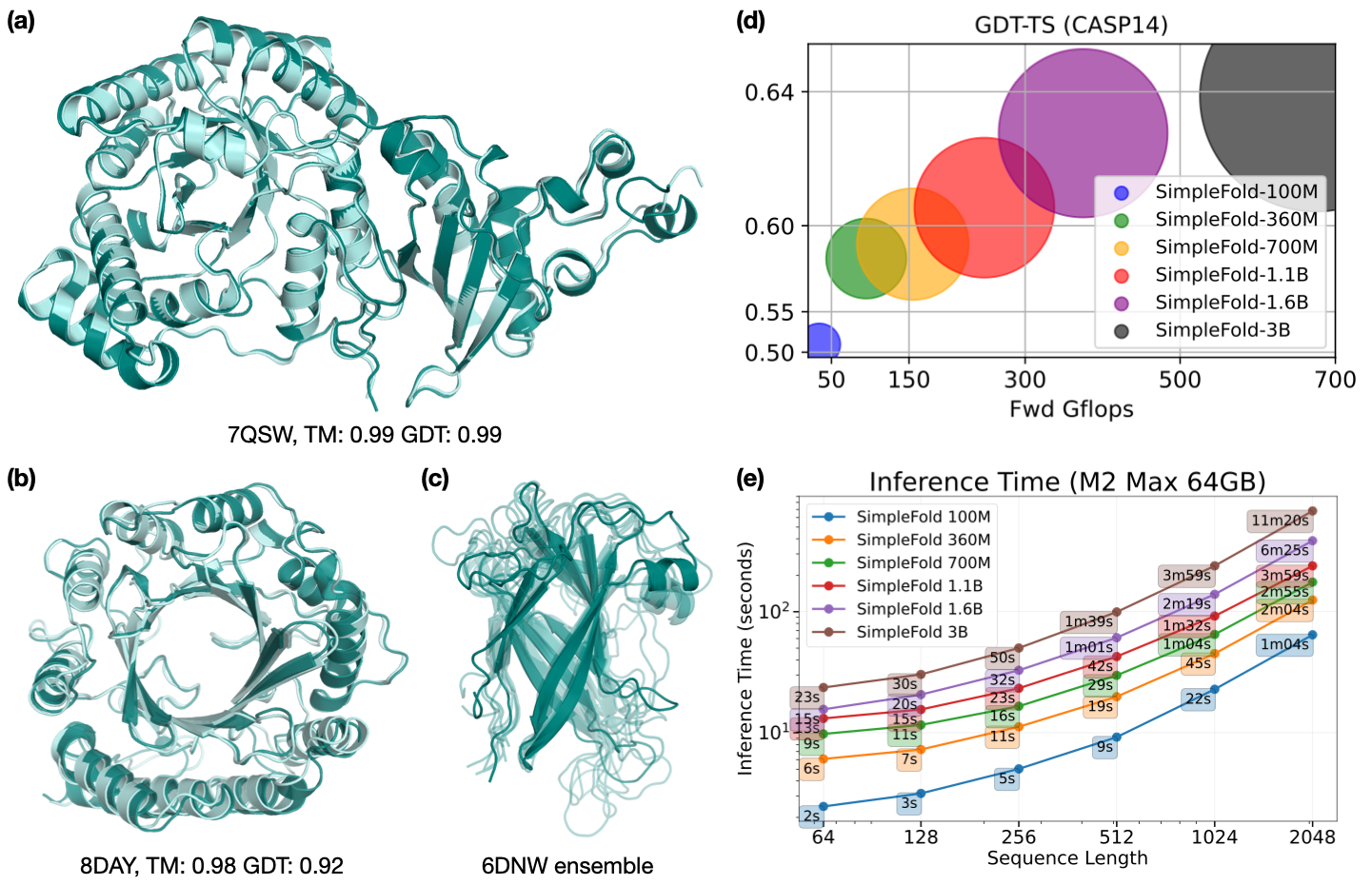
فلسفهای نوین در طراحی مدل
SimpleFold اولین مدل تاخوردگی پروتئین مبتنی بر «تطبیق جریان» (flow-matching) است که کاملاً با استفاده از لایههای ترنسفورمر عمومی ساخته شده است. این مدل از ماژولهای گرانقیمت و مختص-دامنه مانند «توجه مثلثی» (triangle attention) فاصله میگیرد و در عوض، از یک هدف یادگیری مولد مبتنی بر تطبیق جریان بهره میبرد که معماری را ساده کرده و در عین حال عملکردی قدرتمند را حفظ میکند. SimpleFold با مقیاس شگفتانگیز ۳ میلیارد پارامتر، بزرگترین مدل تاخوردگی پروتئین است که تاکنون توسعه یافته و بر روی یک مجموعه داده عظیم شامل بیش از ۸.۶ میلیون ساختار پروتئینی تجربی و استنتاجی آموزش دیده است.
این طراحی ساده نه تنها با پیشرفتهترین مدلهای پایه در بنچمارکهای استاندارد رقابت میکند، بلکه به لطف هدف آموزشی مولد خود، در «پیشبینی گروهی» (ensemble prediction) نیز عملکردی برجسته دارد. SimpleFold یک مسیر جایگزین هیجانانگیز برای پیشرفت در پیشبینی ساختار پروتئین را برجسته میکند و بر مقیاسپذیری و سادگی معماری تأکید دارد.
کار با SimpleFold را آغاز کنید
این پروژه به صورت متنباز منتشر شده و شما میتوانید همین حالا کار با آن را شروع کنید. برای نصب بسته از مخزن گیتهاب آن، دستورات زیر را اجرا کنید:
git clone https://github.com/apple/ml-simplefold.git
cd ml-simplefold
python -m pip install -U pip build; pip install -e .
pip install git+https://github.com/facebookresearch/esm.git # اختیاری برای بکاند MLXپیشبینی ساختارهای پروتئینی
پس از نصب، میتوانید با استفاده از خط فرمان، ساختارها را از فایلهای FASTA خودتان پیشبینی کنید. این مدل از هر دو بکاند PyTorch و MLX پشتیبانی میکند که MLX برای کاربران سختافزارهای اپل توصیه میشود. شما میتوانید از میان اندازههای مختلف مدل، از ۱۰۰ میلیون تا ۳ میلیارد پارامتر، انتخاب کنید.
simplefold \
--simplefold_model simplefold_3B \ # انتخاب مدل از 100M تا 3B
--num_steps 500 --tau 0.01 \
--nsample_per_protein 1 \
--plddt \ # خروجی گرفتن امتیاز اطمینان pLDDT
--fasta_path [YOUR_FASTA_FILE_OR_DIR] \
--output_dir [YOUR_OUTPUT_DIR] \
--backend [mlx, torch]ارزیابی و آموزش مدل شخصیسازیشده
تیم توسعهدهنده، ساختارهای از پیش پیشبینیشده برای چندین بنچمارک کلیدی مانند CAMEO22 و CASP14 را فراهم کرده است که امکان ارزیابی آسان را فراهم میکند. برای کسانی که به دنبال فراتر رفتن از این مرزها هستند، مخزن شامل دستورالعملهای جامعی برای آموزش یا تنظیم دقیق SimpleFold بر روی مجموعه دادههای سفارشی است. فرآیند آموزش از دادههای PDB، AFDB SwissProt و AFESM استفاده میکند و لیست اهداف برای بازتولید تنظیمات اولیه آموزش نیز ارائه شده است.
این دستاورد، امکانات جدیدی را برای پژوهشگران در حوزههای بیولوژی محاسباتی و هوش مصنوعی باز میکند. SimpleFold با سادهسازی معماری هسته، راه را برای مدلهای بزرگتر و تواناتر در آینده هموار میسازد.
اطلاعات بیشتر را کشف کرده و خودتان کدها را بررسی کنید.
منبع: پروژه SimpleFold در گیتهاب
